Les plus récentes avancées dans la recherche et le traitement du cancer ont été réalisées dans le domaine de l'immuno-oncologie, en utilisant de nouvelles immunothérapies pour renforcer le système immunitaire de l'hôte afin de cibler et de détruire plus efficacement les cellules tumorales. Parmi la liste des thérapies à activité immunorégulatrice approuvées par la FDA figurent les immunothérapies à base d'anticorps et de cytokines, les médicaments à petites molécules et les thérapies cellulaires. Le succès que ces thérapies et d'autres ont rencontré en termes de prolongation de la survie est a l'origine de l'accent mis sur le développement de nouvelles thérapies à agent unique et combinaisons thérapeutiques plus efficaces pour traiter le cancer.
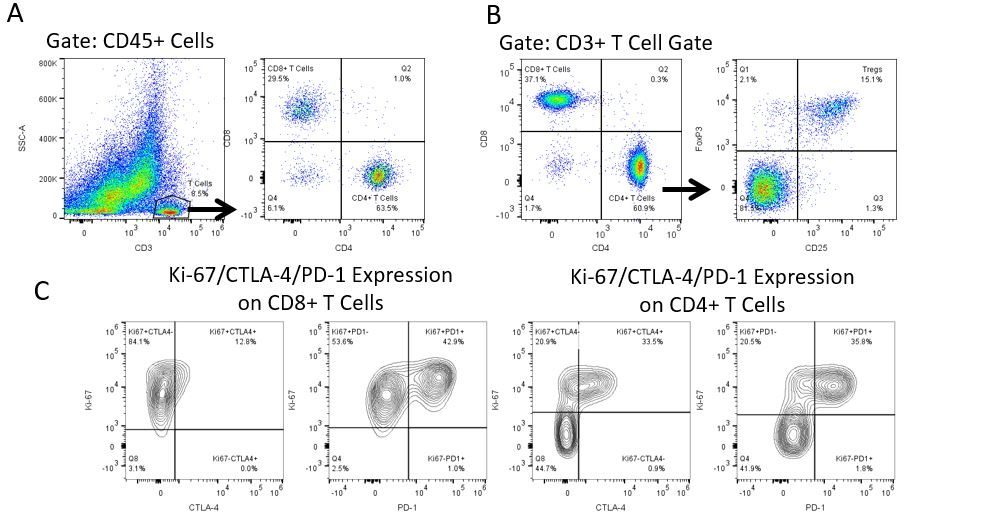
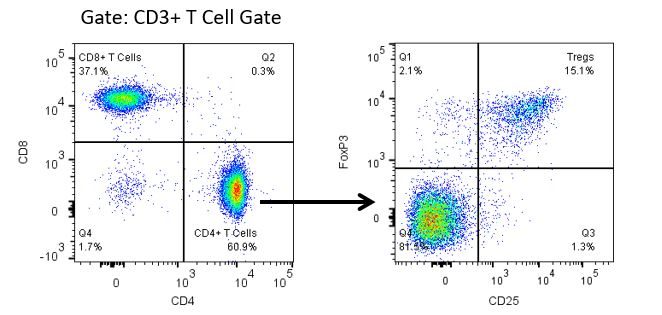
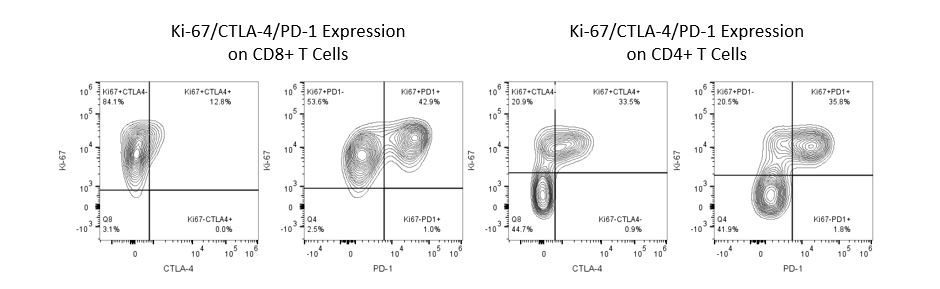
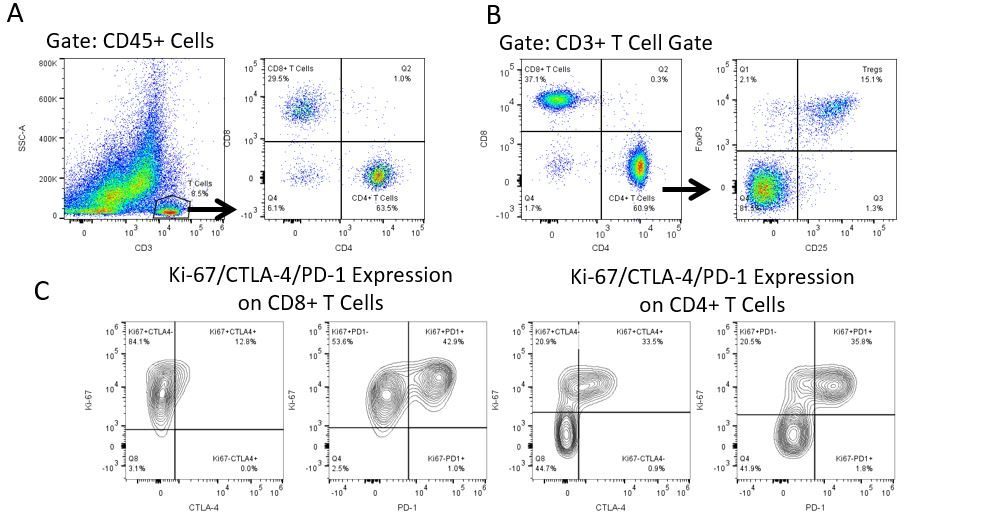
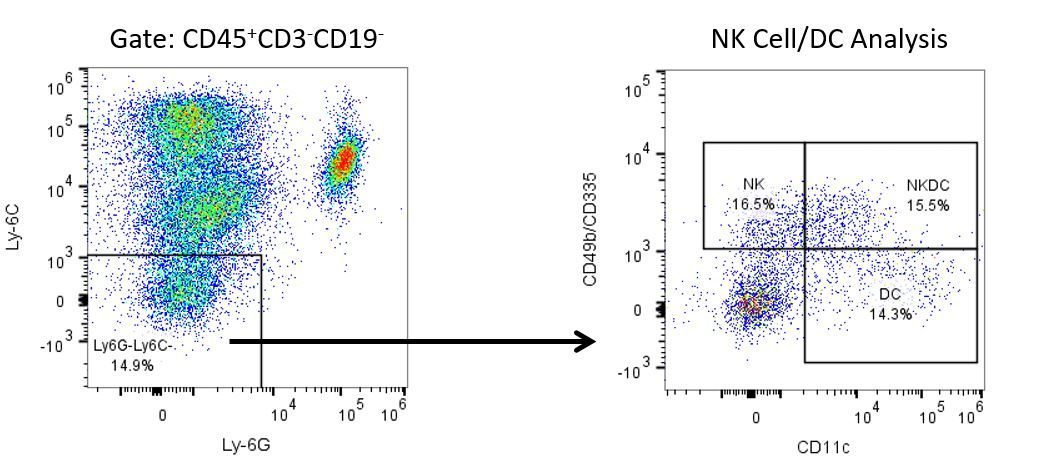
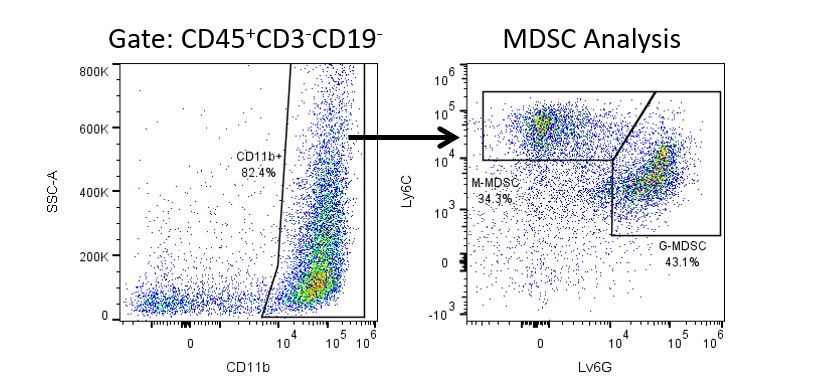
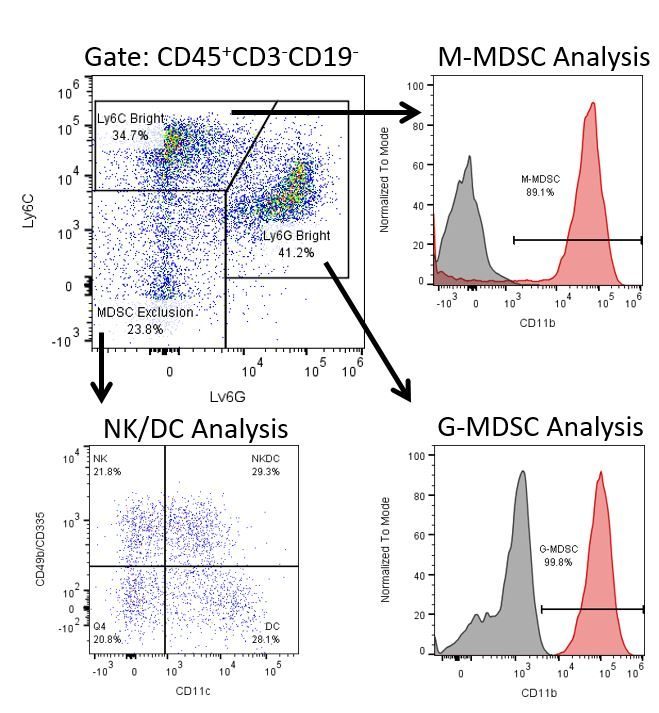
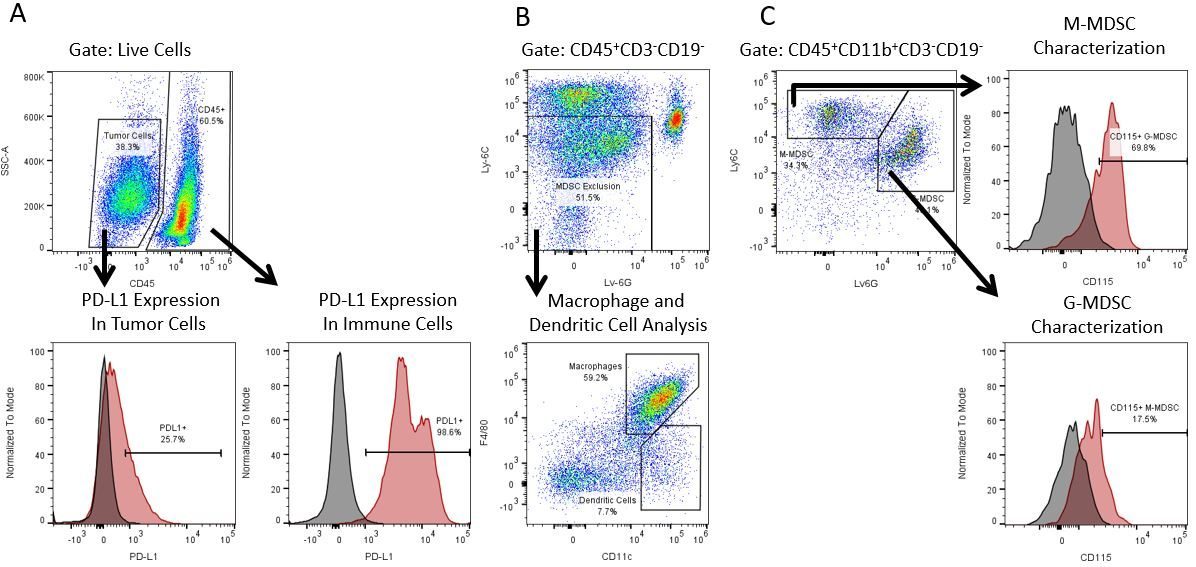
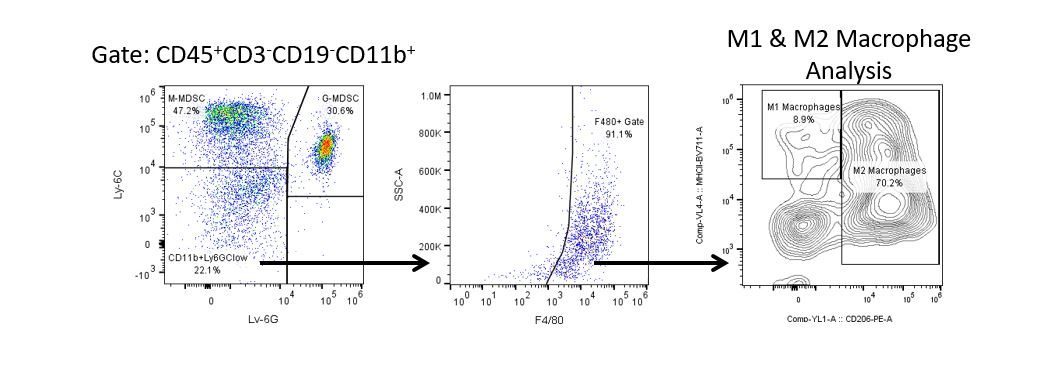
Pour développer de façon productive des immunothérapies, il faut une analyse quantitative à haut débit et robuste du système immunitaire dans le micro-environnement de la tumeur, le sang périphérique et les autres tissus-hôtes.
Pour répondre à ce besoin, notre service de cytométrie en flux sous contrat, vous fournit une ressource de pointe en cytométrie en flux analytique pour vous soutenir dans toutes les facettes de votre développement de médicaments. Faites vos études de génération d'échantillons avec nous ou envoyez-nous sous un jour vos échantillons précliniques ou cliniques non réglementés par la CLIA et nous nous chargerons de la cytométrie en flux pour vous.